2019 年 1 月 31 日 - 作者:Gunjan Baid、Helen Li 和 Pi-Chuan Chang
编者注:本文内容与 Google DeepVariant 博客 上发布的内容相同。
简介 本文将 DNA 测序错误校正描述为一个多类分类问题,并提出了两种深度学习解决方案。我们的第一种方法校正单个读中的错误,而第二种方法(如图 1 所示)从多个读中构建共识,以预测正确的 DNA 序列。我们 Colab 笔记本教程 使用 Nucleus 和 TensorFlow 库实现了第二种方法。我们的目标是展示如何将 Nucleus 与 TensorFlow 结合使用,以解决基因组学中的机器学习问题。


获得更高质量数据集的一种简单方法是丢弃可能包含错误的数据,方法是丢弃整个读或修剪质量低的区域。这种方法并不理想,因为它会导致最终数据集变小。此外,某些序列上下文自然具有更高的错误率,从而导致抽样偏差。因此,存在大量研究致力于开发更复杂的方法来进行错误校正。大多数已开发的方法可以分为两类:
NA12878_sliced.bam - 来自染色体 20(位置 10,000,000-10,100,000)的 Illumina HiSeq 读,下采样到 30x 覆盖率。NA12878_sliced.bam.bai - NA12878_sliced.bam 的索引。NA12878_calls.vcf.gz - 来自 Genome in a Bottle 的 NA12878 的变异真值集。NA12878_calls.vcf.gz.tbi - NA12878_calls.vcf.gz 的索引。hs37d5.fa.gz - hs37d5 的参考基因组。hs37d5.fa.gz.fai 和 hs37d5.fa.gz.gzi - hs37d5.fa.gz 的索引文件。在我们的实现中,我们使用了一个标准的卷积架构,它包含两个卷积层,后面跟着三个全连接层。我们使用非线性 ReLU 层来提高模型的表达能力。卷积层之后的最大池化缩小了输入体积,全连接层之后的 dropout 充当正则化器。注意,我们没有在最后一个全连接层之后包含 softmax 层,因为我们使用的损失函数在内部应用 softmax。每层的详细信息可以在 教程 中找到。 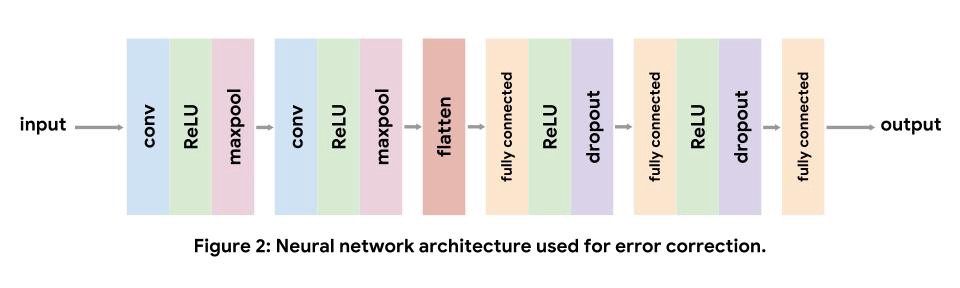
 出于教学目的,我们将问题简化为以下几种情况:
出于教学目的,我们将问题简化为以下几种情况:我们通过将参考基因组分成固定长度的非重叠部分来生成输入序列。在训练、评估和测试时,我们通过将参考序列中的碱基归零来模拟缺失的碱基,如图 3 所示(位置 5)。除了使用参考基因组来模拟缺失数据之外,我们还可以将这种模型应用于测序读中的数据,特别是质量分数低于阈值值的碱基。
 出于教学目的,我们将问题简化为以下几种情况:
出于教学目的,我们将问题简化为以下几种情况: 与第一种方法不同,我们没有使用参考基因组来训练该模型。相反,我们的训练数据来自映射的 Illumina HiSeq 读。该网络的输入是映射读中观察到的归一化碱基计数矩阵,以我们想要预测的正确碱基的位置为中心。 Clairvoyante(用于变异调用的神经网络)的作者以及 Jason Chin 的示例方法 中使用了类似的特征化。网络的输出是所有可能碱基的分布,最终预测是概率最高的碱基。与第一种方法类似,标签集使用参考基因组中观察到的碱基生成。我们使用包含错误的示例(堆积中至少有一个读与中心位置的参考不匹配)和不包含错误的示例(堆积中的所有读都与中心位置的参考匹配)的混合示例。
与第一种方法不同,我们没有使用参考基因组来训练该模型。相反,我们的训练数据来自映射的 Illumina HiSeq 读。该网络的输入是映射读中观察到的归一化碱基计数矩阵,以我们想要预测的正确碱基的位置为中心。 Clairvoyante(用于变异调用的神经网络)的作者以及 Jason Chin 的示例方法 中使用了类似的特征化。网络的输出是所有可能碱基的分布,最终预测是概率最高的碱基。与第一种方法类似,标签集使用参考基因组中观察到的碱基生成。我们使用包含错误的示例(堆积中至少有一个读与中心位置的参考不匹配)和不包含错误的示例(堆积中的所有读都与中心位置的参考匹配)的混合示例。 


2019 年 1 月 31 日 — 发布者 Gunjan Baid、Helen Li 和 Pi-Chuan Chang
编者注:这篇文章与 Google DeepVariant 博客 上发布的内容相同。
引言在本篇文章中,我们将 DNA 测序错误校正问题表述为一个多类别分类问题,并提出两种深度学习解决方案。我们的第一种方法在单次读取中纠正错误,而第二种方法(如图 1 所示)…